中文标题:生物医学领域的预训练模型BioBERT
英文标题:BioBERT: a pre-trained biomedical language representation model for biomedical text mining.
发布平台:Bioinformatics
发布日期:2020-02-15
引用量(非实时):3200
DOI:10.1093/bioinformatics/btz682
作者:Jinhyuk Lee, Wonjin Yoon, Sungdong Kim, Donghyeon Kim, Sunkyu Kim, Chan Ho So, Jaewoo Kang
文章类型:journalArticle
品读时间:2022-03-10 17:26
1 文章萃取
1.1 核心观点
通用领域预训练模型与生物医药领域预训练模型存在词分布不一致的问题;基于此,本文借助BERT模型与生物医药领域预料,训练微调并开源了BioBERT模型,并在生物医药领域的命名实体识别、关系提取和问题回答(NER、RE和QA)等任务上取得了非常优异的效果。
1.2 综合评价
- BERT在生物医药细分领域的版本
- 有相关开源代码与预训练模型
- 结果经过多方验证,可靠有效
1.3 主观评分:⭐⭐⭐⭐
2 精读笔记
2.1 背景与方法
BERT:一种从大量无标注文本中学习单词表示的双向编码器。借助Transform结构和随机掩盖单词的学习方法,BERT在NLP各个领域都取得了非常优秀的效果。
BioBERT:基于通用文本(英文维基2.5B、图书语料库0.8B)训练出的BERT,无法处理医疗领域大量专业名词和术语的吃力,因此本文基于医疗领域文本(PubMed摘要 4.5B、PMC文章全文 13.5B)对BERT进行微调,最终得到了BioBERT。
WordPiece:BERT用于处理单词的方法,借助BPE(Byte-Pair Encoding)双字节编码的方法对单词进行再拆分以方便模型更好地进行语言的表达与编码(比如"loved"可能会被拆分为"lov"和“ed”,而"loving"可能会被拆分为"lov"和“ing”,从而方便模型进行语义与时态的区分,具体拆分方法参考)。为了方便BioBERT与BERT间的转换,BioBERT使用与BERT一致的WordPiece。
BioBERT的评测主要围绕命名实体识别、关系提取和问题回答这三个方面
NER:命名实体识别(Named entity recognition),本文中采用的标注方法为BIO2,评价方法为召准率 precision、召回率 recall 和F值。
RE:关系提取(Relation extraction),其实就是对已识别实体的分类,模型预测类别的输出用[CLS]标识符进行占位,标注形式举例:@DISEASE$,评价方法为召准率 precision、召回率 recall 和F值。
QA:问题回答(Question answering),验证数据集使用BioASQ factoid,并排除其中无法回答的问题样本,评价方法为strict accuracy(回答是完全正确的), lenient accuracy(前五种回答存在正确答案的概率) and mean reciprocal rank(正确答案排名的平均值)
训练过程:针对$BERT_{LARGE}$的预训练,本文使用8个V100(32 GB)GPU进行微调。最大序列长度为512,mini-batch size设置为192,预训练BioBERT v1.0 耗时10多天( + PubMed + PMC),预训练BioBERT V1.1 (+ PubMed) 耗时近23天;针对$BERT_{BASE}$的预训练,本文使用Titan XP(12 GB)GPU进行微调,时间上快了很多,RE和QA任务耗时不到一小时,NER任务运行了大概20多个epochs。
2.2 评价与结论
NER任务:
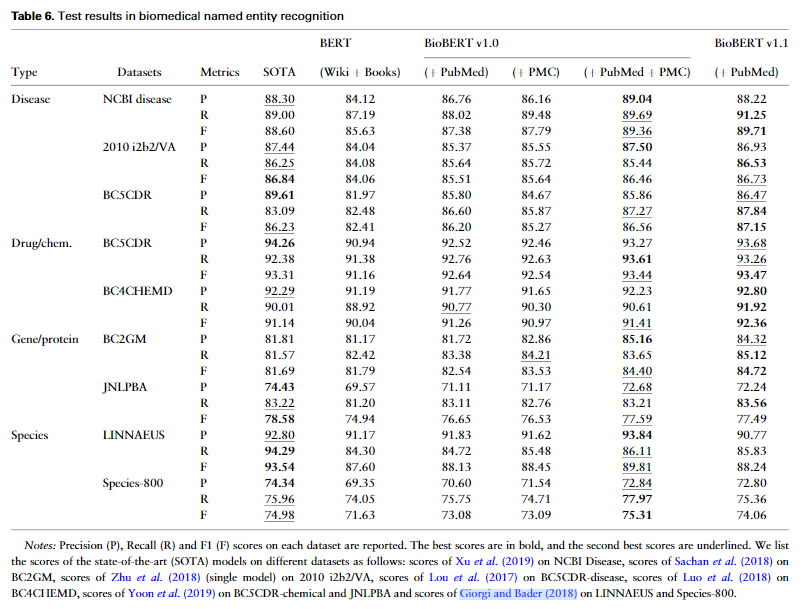
以F值为评估指标,9份数据集中,BioBERT在其中7份取得最优(超过SOTA)的结果
RE任务:
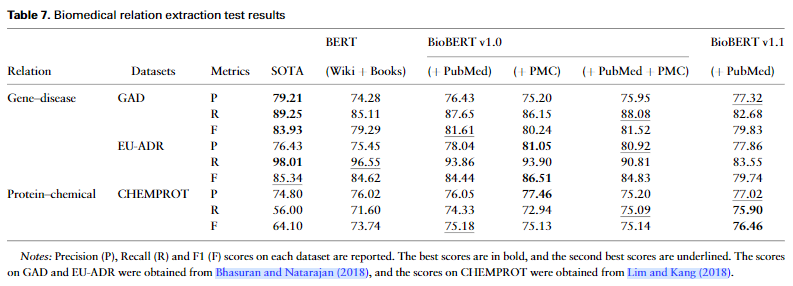
以F值为评估指标,3份数据集中,BioBERT在其中2份取得最优(超过SOTA)的结果
QA任务:
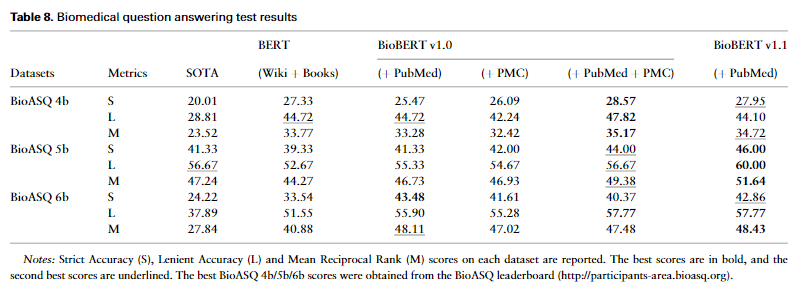
3份数据集中,每份数据集有3种评价指标,共9个指标,,BioBERT在其中7份取得最优(超过SOTA)的结果
性能表现VS词表大小:
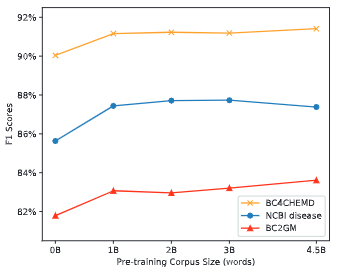
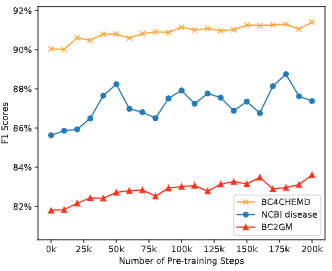
性能表现VS训练次数:
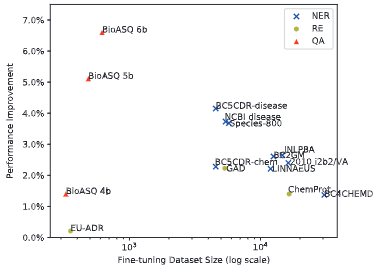
性能提升VS不同数据集:
相关资源
- 在线论文
- 预训练模型
- 模型源码
- 本地文件地址:2020_BioBERT_Lee et al.pdf
- 本地Zotero地址:2020_BioBERT_Lee et al.pdf